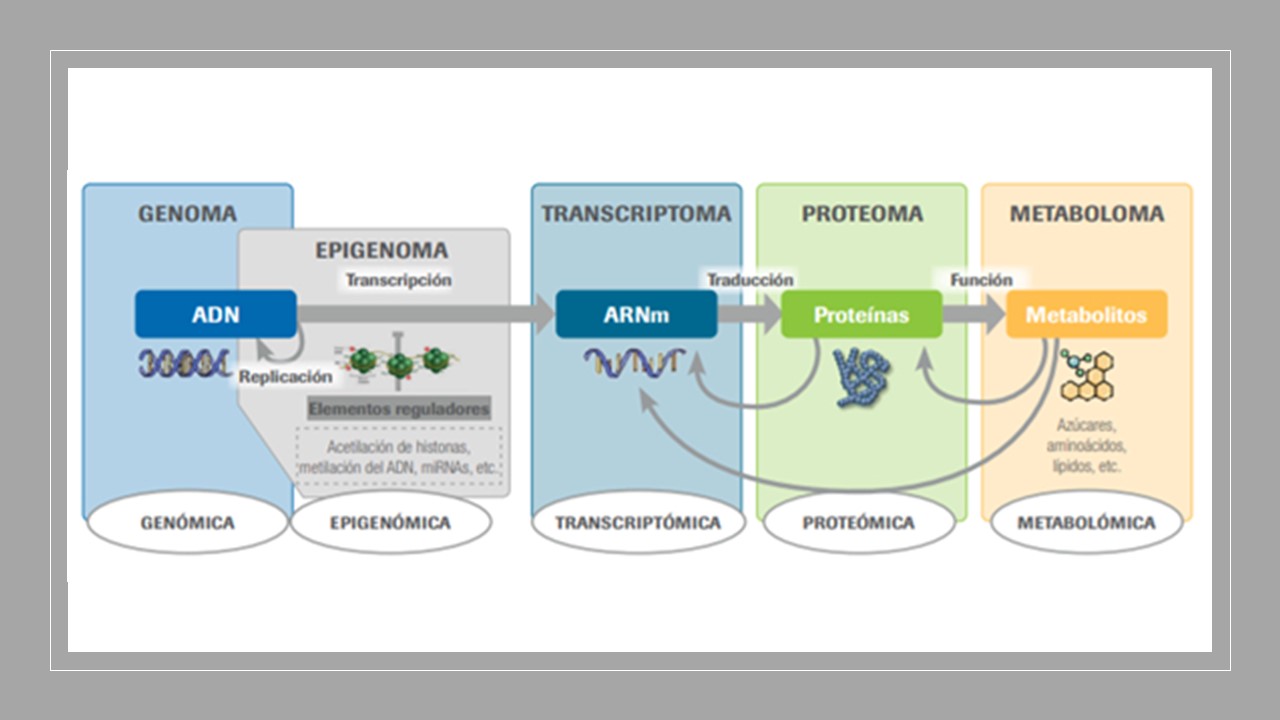
Sarrera honetan, “omikak” NEIKEReko Animalien Osasun Sailean garatzen ari garen proiektuetan nola txertatu ditugun kontatzen jarraituko dugu. Kasu honetan, paratuberkulosiari (PTB) gutxiago eragiten dioten eta gehiago irauten duten animaliak aukeratzen laguntzeko eta gaixotasuna goiz detektatzeko metodo diagnostiko berriak diseinatzen laguntzeko, genomikak, transkriptomikak eta proteomikak informazioa nola eman dezaketen esango dizuegu bi proiekturen bidez:
PARACON proiektuan (RTA2018-094192-R-C01) behi-biomarkatzaileak (mRNAak, miRNAak eta proteinak) identifikatzeko transkriptomika eta proteomika erabili ziren. Biomarkatzaile horiei esker, infekzioaren patogenesia hobeto ezagutu ahal izan da eta infekzioaren estadio goiztiarretan kutsatutako animaliak, gorozkietan Mycobacterium avium subsp paratuberculosis (MAP) kanporatzen edo zeinu klinikoekin hasi gabekoak, hautemateko diagnostiko-metodo berriak garatzeko oinarriak ezarri ahal izan dira. Biomarkatzaile berrien diagnostikorako gaitasuna MAPek modu naturalean kutsatutako eta infekzioaren estadio desberdinetan zeuden animalien laginak erabiliz balioztatu da. Bestalde, proiektu honetan MAP bidezko kutsadurarekiko sentikortasun txikiagoa duten animalien hautapen genetikoan lan egin da, hau modu zabalean onartutako kontrol-estrategia da. Horretarako, PTBrekiko suszeptibilitate genetikoaren desberdintasunei lotutako sekuentzia bakarreko polimorfismoak (SNPak) bilatzeko analisi genomikoak egin ziren. Helburu hori lortzeko, Frisona Españolako Elkarteen Konfederazioarekin (CONAFE) lankidetzan jardun zen. Konfederazio hori Eurogenomics-eko kidea da eta europar kooperatiba honek, europako behi-populazioaren hautapen genetikorako EUROG10K txipa garatu zuen.
PTBari erresistentzia eta tolerantzia ematen dioten eskualde genetikoen ezagutza mugatua da, baita erresilientzia-moten kontrolean parte hartzen duten mekanismo molekularren eta zelularren ezagutza ere. PARARESILIENCE (PID2021-122197OR-C21) proiektuan, hurbilketa berriak landuko dira, hala nola transkriptomikaren eta proteomikaren bitartez, biomarkatzaileak (mRNAak eta proteinak) aurkituz. Biomarkatzaile berri hauek entsegu berrietan erabiliko dira, non helburua osasuna eta ekoizpena kalteturik ez duten eta gaixotasuna jasateko eta/edo kutsadurari eusteko animalia asintomatikoen identifikazioa izango da. Gaixotasuna kontrolatzeko eta baita hautapen genetikorako ere, biomarkatzaile horien mailak neurtzea oso baliagarria izan liteke. Testuinguru horretan, PTBrekiko erresilientzia-biomarkatzaileak azkar eta zehatz atzemateko, “point of care” (POC) plataforma garatu nahi da. Bestalde, osasun-karaktereen ebaluazio genetikoen inplementazioak izan duen erronka handienetako bat, modu zehatzean, errepikakorrean eta laborategien artean konparatzeko moduan, animalia kopuru nahiko baten neurri fenotipikoak lortzeko zailtasuna izan da. Neurri fenotipiko horiek, genomikoki hautematen dena animalien zeluletan adierazten edo ikusgai dagoela erakusten duen froga bat eskatzen dute. Proiektu honetan, fluoreszentziako mikroskopia denbora errealean erabiliz, makrofagoetan MAPen hazkuntza neurtzeko protokolo bat estandarizatuko da, etorkizunean aukeraketa genetikoko planetan erabili ahal izateko. Makrofagoetan MAP bidezko infekzioa modu eraginkorragoan (erresistenteagoan) mugatzeko gai diren animaliek, estres gutxiago izango dute eta ez dute ekoizpena eta osasuna konprometiturik izango, bizitza-luzeran eragin positiboa izango duelarik. Halaber, hurbilketa genomiko-transkriptomiko konbinatu baten bidez, PTBrako erresilientzian paper funtzionala duten geneen adierazpena erregulatzen duten eskualdeak (cis-eQTLak) mapeatu eta aztertuko dira. Gene-aldagai funtzional horiek hautapen genetikoko programetan sartzeak, MAP bidezko infekziorako erresilientzia nabarmen areagotzea espero da.